Regulation der Sumoylierung durch die Sumoylierung des E2-Enzyms (Ubc9)
Labor Pichler
Die Sumoylierung wird primär über E3-Ligasen und SUMO-spezifische Proteasen reguliert, da diese Enzyme hauptsächlich die Substratspezifität sicherstellen. Wir haben einen alternativen Mechanismus der Regulation auf der E2-Ebene charakterisiert: Die Sumoylierung des E2-Enzyms (Ubc9). Diese Modifikation ist von der Hefe bis zu den Säugetieren konserviert, nutzt aber strukturell unterschiedliche Modifikationsstellen, was auf unterschiedliche enzymatische Konsequenzen schließen lässt.
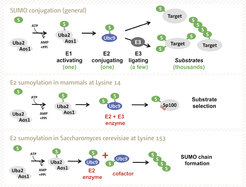
In Säugetierzellen fanden wir, dass die N-terminale Ubc9-Sumoylierung die Affinität und Modifikation ausgewählter Substrate in Abhängigkeit von einem nicht-kovalenten SUMO-Interaktionsmotiv (SIM) erhöht (Knipscheer, Klug et al., Mol Cell 2008, Abbildung 2, mittlere Tafel).
Im Gegensatz dazu führt die C-terminale Ubc9-Sumoylierung in der Hefe Saccharomyces cerevisiae zur E2-Inaktivierung, aber verwandelt das inaktive Ubc9*SUMO zu einem Kofaktor für das unmodifizierte Ubc9. Zusammen kooperieren diese Enzymvarianten bei der SUMO-Kettenbildung, die für die Ausbildung des synaptonemalen Komplexes (SC) in der Hefe-Meiose wichtig ist (Klug et al, Mol Cell 2013, unteres Panel).
Derzeit fokussieren wir uns auf neue Substrate, die wir für das sumoylierte Ubc9 in Säugerzellen identifiziert haben, und untersuchen wie die Sumoylierung ihre biologischen Funktionen in Abhängigkeit vom sumoyliertem Ubc9 und SUMO E3-Ligasen reguliert.
